Regeringer, videnskabsmænd og forskningsinstitutioner bruger teknologi og innovation til at tackle COVID-19.
Open source-værktøjer er blevet udviklet, og nogle bliver fortsat testet for at understøtte de bestræbelser, der er blevet implementeret for at minimere resultaterne af denne dødelige virus.
En række open source-værktøjer er allerede på plads, som hjælper forskere med at lære om sygdommen, identificere spredningen, forhindre spredningen og minimere yderligere spredning og dødsfald. En række projekter er tilgængelige på Uddannelses økosystem der beskriver, hvordan man bruger forskellige værktøjer.
Denne artikel ser på nogle af de open source-værktøjer og -biblioteker, der bliver brugt til overvågning, forebyggelse og indeslutning, diagnosticering og behandling af COVID-19.
Indholdsfortegnelse
COVID-19 sporingskort
I januar lancerede JHU’s Center for Systemvidenskab og Engineering en COVID-19 Global Tracking Map som hurtigt blev en internationalt betroet kilde til den udviklende pandemis data i realtid.
Kortet er open source og er blevet brugt til at drive forskning og visualiseringsindsats af topmedieorganisationer, små organisationer og lokale myndigheder.
DXY-COVID-19-Crawler
DXY-COVID-19-Crawler er et af de tidligste open source-projekter udviklet til at reagere på COVID-19 i januar.
Udviklerne gjorde brug af data fra DXY.cn, et websted, der blev brugt af kinesiske læger til at rapportere og spore tilfælde. De udviklede en webcrawler, der indsamlede data fra webstedet og gjorde det tilgængeligt via en API og et datavarehus. Koden er tilgængelig på Github.
Åbn Data Kit
Open Data Kit (ODK) er ikke et nyt værktøj. Det er blevet brugt før under ebola-udbruddene i Vestafrika i 2004 og det nyere udbrud i Den Demokratiske Republik Congo i 2019.
Det hjælper hovedsageligt brugere med at indsamle, administrere og bruge data i kontaktsporing, strategisk kortlægning, beslutningsstøtte, samfundssensibilisering og sagsbehandling.
ODK-fællesskabet har videreudviklet sin brug som reaktion på COVID-19 ved at stille en rapporteringsformular til rådighed i sine implementeringer. Skemaet er udformet efter Verdenssundhedsorganisationens protokol til undersøgelse af sager og kontaktopsporing og undersøgelser.
De ledende udviklere tilbyder også gratis support til enhver organisation, der har implementeret ODK som reaktion på COVID-19.
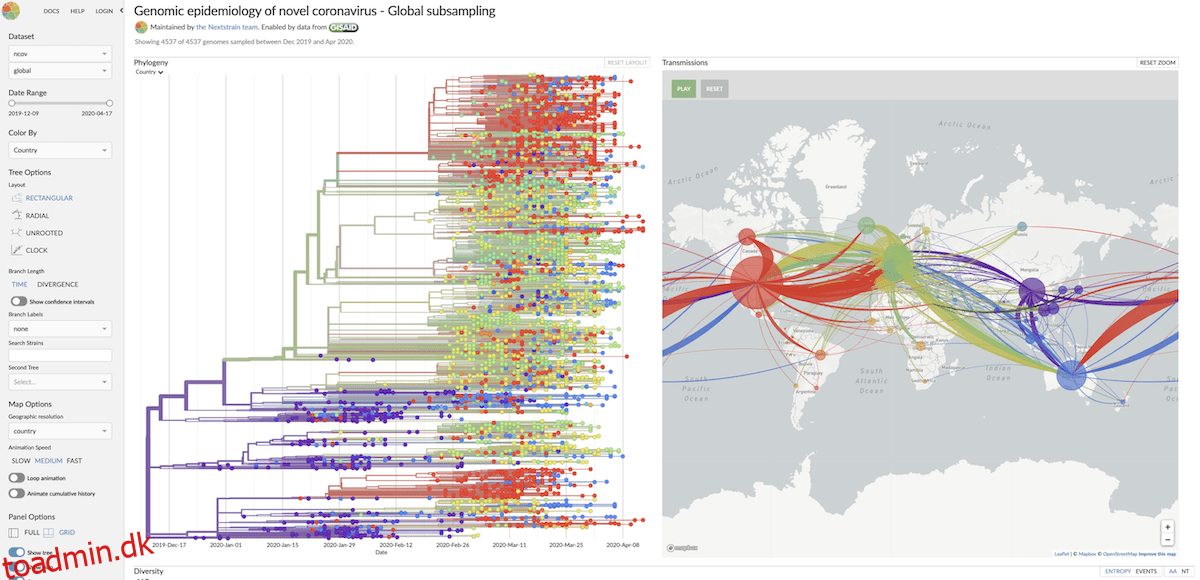
Nextstrain
Nextstrain sporer udviklingen af patogener. Det er tidligere blevet brugt til at beregne en sygdoms familiehistorie, hvilket muliggør forudsigelse af sygdomsforløbet.
Det er med succes blevet brugt i tidligere epidemier, såsom ebola. Gennem genetiske data fra Global Initiative on Sharing All Influenza Data (Gisaid) bliver Nextxtrain brugt til at tackle COVID-19.
DHIS2
Du ved det sikkert allerede DHIS2. Det er verdens største Health Information Management System. Det bruges i mere end 70 lande. Som en del af reaktionen på COVID-19 har DHIS2 udgivet en digital datapakke, der fremskynder infektionsdetektion, rapportering, overvågning og behandling af sygdommen.

Den digitale DHIS2-datapakke gør brug af standardmetadata, der er tilpasset WHO’s protokol om COVID-19-tilfældedefinition og overvågning for at muliggøre hurtig implementering og respons.
Pikobar West Java
Indonesiens informations- og koordinationscenter for sygdom og katastrofer er et kriseberedskabscenter, der er dannet for at afbøde og reagere på COVID-19 i Vest-Java-provinsen i Indonesien.
Jabar Digital Service har som en del af svaret udviklet en open source webværktøj og app der giver brugerne adgang til de seneste COVID-19-data.
OpenMRS
OpenMRS er et patientplejesystem, der bruges i mange udviklingslande over hele kloden. På grund af fleksibiliteten i OpenMRS systemkan lande, der har haft deres sundhedsressourcer anstrengt, bruge det til overvågning, screening og behandling af COVID-19.
Systemet kan hjælpe dem med at udvide deres kapacitet ved at give dem adgang til videnskabeligt baseret information om håndtering af krisen.
OpenLMIS
Det OpenLMIS projekt gør brug af en samfundsfokuseret taktik til at udvikle et open source og et justerbart logistikstyringsinformationssystem. OpenLMIS-systemet søger at forbedre datanøjagtigheden, øge ansvarligheden, forbedre dataens aktualitet og synlighed.
OpenLMIS-systemet søger at forbedre sundhedsforsyningskæderne, opgørelsen af medicinske ressourcer for at give et klart billede af de tilgængelige medicinske forsyninger, herunder testsæt og PPE’er (personlige værnemidler). Dette værktøj kan effektivt bruges til at støtte beslutningstagere i at allokere ressourcer som en reaktion på COVID-19.
Sundhedssider
Det Global Healthsites Mapping Project er et projekt, der har til formål at kortlægge alle sundhedsfaciliteter i verden og gøre detaljerne på hvert hospital let tilgængelige. Data om sundhedsfaciliteter er gjort tilgængelige via en API.
Ved at samarbejde med brugere fanger og validerer healthsites.io-teamet placeringen og kontaktoplysningerne for hver sundhedsfacilitet og gør dataene frit tilgængelige og tilgængelige via en Open Data License.
SORMAS
SORMAS (Overvågningsudbrud Response Management og Analyse System) er et open source mobilt e-sundhedssystem. Det er blevet indsat til at implementere sygdomsbekæmpelse samt udbrudshåndteringsprocedurer.
Det er blevet implementeret effektivt i flere lande, herunder Ghana, Nigeria, Nepal og Fiji, til COVID-19-overvågning og tidlig opdagelse.
SORMAS er et gratis, open source-system, der følger databeskyttelsesstandarder.
Tokyo COVID-19
I modsætning til mange andre byer og regeringer Tokyo Metropolitan Government har udviklet et open source-websted, der informerer sine beboere om COVID-19. Ved at gøre det open source har siden set bidrag fra over 200 brugere. Tre andre byer, Chiba, Nagano og Fukuoka, har genskabt hjemmesiden.
OpenELIS
Formålet med OpenELIS sundhedssystem er at forbedre sundhedsvæsenet ved at levere et progressivt, standardbaseret laboratorieinformationsstyringssystem, der kan bruges af forskellige sundhedsinitiativer til at forbedre behandlingsmulighederne.
COVID-19-udbruddet har givet en global udfordring med kontaktsporing og massetest af formodede tilfælde. OpenELIS-systemet kan effektivt implementeres til at tackle COVID-19 for at lette sporing af laboratorietests og resultater.
Det Community Health Toolkit er en samling af open source-værktøjer samt open access-ressourcer, der sigter mod at bygge og implementere digitale værktøjer til brug i lokale sundhedsinitiativer på områder, der er svære at nå.
Community Health Toolkit-udviklerfællesskabet har mobiliseret sig for at udvikle værktøjer og ressourcer, der har til formål at støtte sundhedspersonale i lokalsamfundet med at tackle COVID-19.
KLOKKE
Det COVID-19 Hospital Impact Model for Epidemier (CHIME) er en open source-applikation udviklet af dataforskere ved Penn Medicine – University of Pennsylvania. Det er et onlineværktøj, der gør det muligt for hospitaler at forudsige virussens indvirkning på sundhedsressourcerne.
Det er udviklet ved hjælp af Python og pandas open source-afhængighed.
COVID-plejekort
Det COVID-plejekort hjælper med at kortlægge de allerede eksisterende sundhedsressourcer og med at forudsige huller i hospitalssenge, ventilatorer, medicinske forsyninger og personale. Alle metoder, databehandlingsværktøjer, visualiseringer og kildekode er gratis og open source.
COVID Care Map-projektet søger at foregribe og handle for at tilkalde støtte til effektivt at tage sig af det hurtigt voksende antal COVID-19-infektioner og dem, der har behov for intensiv pleje.
Locale.ai
Locale.ai har udviklet en open source, interaktiv visualisering af alle bekræftede tilfælde af COVID-19 over hele kloden. Det stiller spørgsmål til open source-datasættet fra John Hopkins University.
Locale.ai udviklede COVID-19 visualiseringswebsted ved brug af Vue.js, som er en populær ramme, der giver udviklere mulighed for at skabe moderne webapps.
COVID-19 over hele verden
Denne app gør brug af en kortvisualisering til at overvåge spredningen af COVID-19, de bekræftede tilfælde og udviklingen af sygdommen over hele kloden. Det gør brug af data fra John Hopkins CSSE.
Coronavirus tracker
Dette er en skinnende app udviklet af John Coene. Den sporer spredningen af COVID-19 ved brug af data fra John Hopkins, DXY-data og Weixin. Appen viser antallet af mistænkte, bekræftede og genoprettede tilfælde efter tid og region. Koden er tilgængelig på Github.
COVID-19 globale tilfælde
COVID-19 globale tilfælde er en skinnende app udviklet af Christoph Schoenenberger, der viser udviklingen af COVID-19 på et kort, plots, oversigtstabeller og figurer. Dens kode er tilgængelig på Github.
Regeringer og COVID-19
Dette er en Shiny app udviklet af Sebastian Engel-Wolf. Den kortlægger den eksponentielle COVID-19-vækst, dage til fordobling af infektioner, bekræftede tilfælde, dødelighed og antallet af bekræftede tilfælde pr. 100.000 mennesker i forskellige regioner. Koden er tilgængelig på Github.
Afslutter
Open source-fællesskabet har reageret hurtigt og effektivt på COVID-19-pandemien. Mange projekter er blevet bygget og bliver fortsat bygget for at tackle spredningen af sygdommen. Denne artikel beskriver nogle af projekterne. Der er stadig usikkerhed om, hvordan sygdommen udvikler sig i de kommende uger. Flere projekter, der kan udnytte de eksisterende open source-teknologier, vil finde en plads i kampen mod denne dødelige sygdom.
Pas på dig selv!
Artikel af Dr. Michael J. Garbade, CEO, Education Ecosystem